González-Bello
Líneas de investigación
Antibacterianos: Aproximaciones innovadoras para enfrentar el desafío de las súper-bacterias. Dianas terapéuticas inexploradas. Fármacos con Nuevos Modos de Acción. Nuevos kits de diagnóstico
Investigador(es) principal(es)
Miembros del grupo
| Maneiro Rey, María |
Inv. Postdoctoral |
|
| Canabal Facón, Rafael |
Inv. Predoctoral |
|
| Casabella Amieiro, Braulio |
Inv. Predoctoral |
|
| Outeiral Valiño, María Carmen |
Inv. Predoctoral |
|
| Suárez de Cepeda Fuentes, Pilar |
Inv. Predoctoral |
Investigación
Estrategias para Desbloquear la Resistencia Bacteriana
La estrategia actual en el descubrimiento de fármacos para el tratamiento de las enfermedades infecciosas por parte de la industria farmacéutica, no ha cambiado significativamente en los últimos 60 años. Esta implica principalmente modificaciones de medicamentos anteriores que inciden sobre un pequeño número de funciones bacterianas y del mismo tipo, para hacerlos más eficaces y resistentes contra las bacterias resistentes. Sin embargo, la resistencia bacteriana a estos objetivos biológicos es generalizada y bien conocida. Por lo tanto, se necesita con urgencia una nueva estrategia en el descubrimiento de antibióticos para controlar el creciente desarrollo y la propagación de las SÚPERBACTERIAS, que son patógenos que incluso se han vuelto resistentes a múltiples tipos de antibióticos. Nuestro grupo de investigación se centra en el desarrollo de nuevos agentes antibacterianos con mecanismos de acción INNOVADORES para tratar infecciones mortales causadas por los patógenos Gram-negativos multirresistentes, en especial para el tratamiento de las infecciones causadas por las bacterias prioritarias recientemente identificadas por la OMS, es decir, Acinetobacter baumannii, Pseudomonas aeruginosa y Enterobacteriaceae. Estos patógenos son muy peligrosos en pacientes con un sistema inmunitario débil sometidos a quimioterapia contra el cáncer, cirugía general, trasplantes de órganos y diálisis, entre otros, para quienes nuestra capacidad de tratar las infecciones secundarias es crucial. Además, estamos estudiando el potencial de dianas bacterianas inexploradas para el desarrollo de nuevos agentes contra la tuberculosis, enfermedad que se la considera una prioridad de salud mundial.
Actualmente en nuestro grupo de investigación se exploran tres estrategias principales que inciden sobre la VIABILIDAD BACTERIANA, la RESISTENCIA BACTERIANA y la COMUNICACIÓN ENTRE BACTERIAS.
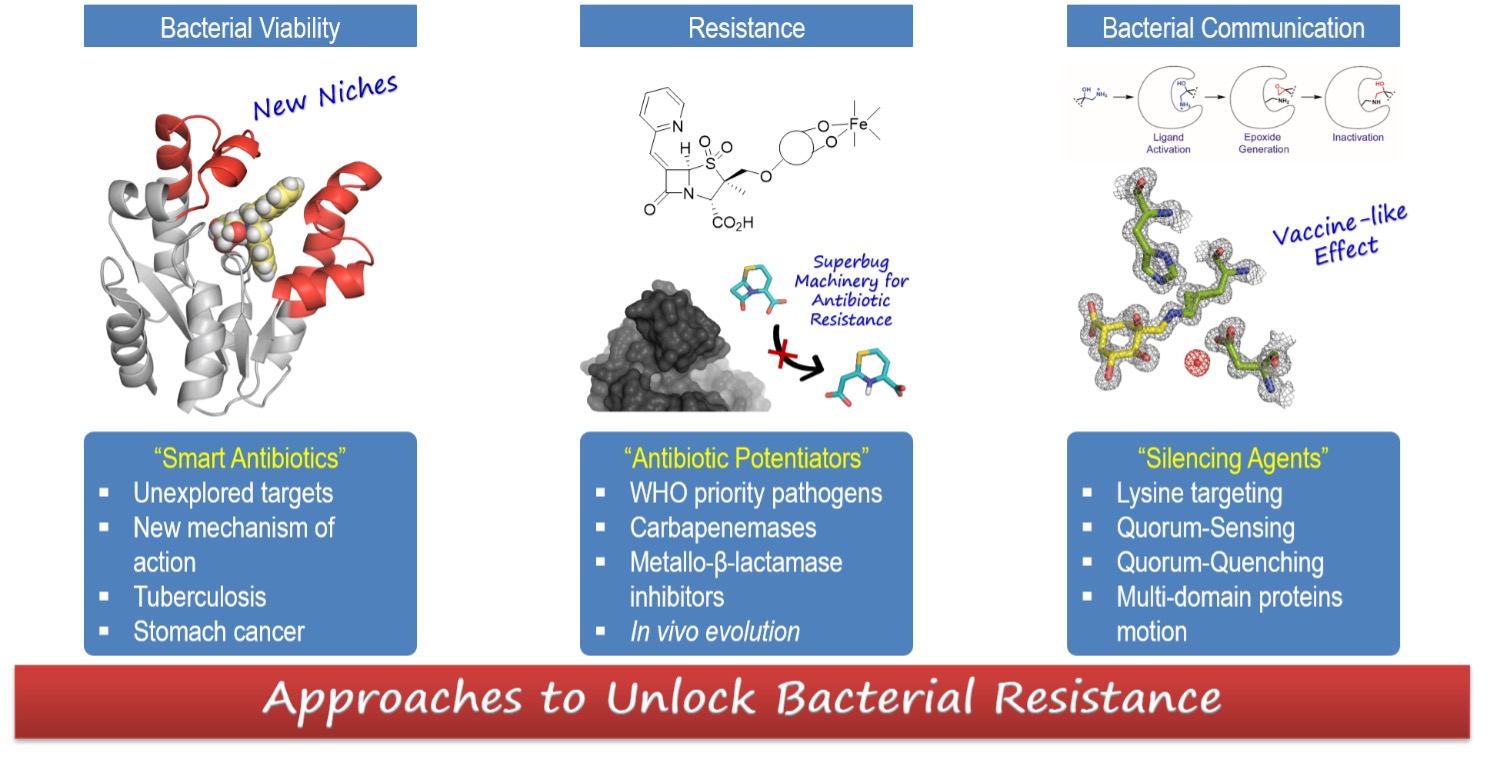
ANTIBIOTICOS INTELIGENTES – Estamos estudiando el potencial terapéutico de varios enzimas de la ruta del ácido siquímico (siquimato quinasa, EPSP sintasa) para el descubrimiento de nuevos antibióticos. Estas dianas terapéuticas poco exploradas son esenciales para la viabilidad de la bacteria. En base al movimiento esencial del enzima para la regeneración catalítica (diseño basado en el movimiento), hemos identificado potentes inhibidores competitivos reversibles que estabilizan una conformación abierta inactiva para la catálisis (J. Med. Chem. 2016, 59, 5471, Chem. Eur. J. 2016, 22, 17988; Org. Chem. Front. 2019, 6, 2514). También hemos desarrollado inhibidores de estos enzimas mediante la rigidificación de la conformación activa de alta energía que reconoce el enzima para la catálisis (J. Am. Chem. Soc. 2013, 135, 12366). Utilizamos un enfoque pro-fármaco para la internalización de los mismos en la bacteria.
POTENCIADORES DE LOS ANTIBIOTICOS – Estamos trabajando en el desarrollo de nuevos antibióticos adyuvantes capaces de bloquear el mecanismo más relevante de la resistencia bacteriana: la inactivación enzimática del antibiótico (β-lactamas) mediante la acción de las β-lactamasas. Desde el punto de vista del descubrimiento de fármacos, esta terapia combinada (antibiótico + antibiótico adyuvante) tiene la ventaja de que no es necesario invertir esfuerzos en la tan costosa y compleja identificación de nuevas dianas terapéuticas. En concreto, nos centramos en las β-lactamasas más peligrosas, las carbapenemasas de clase D (OXA-23, OXA-24/40, OXA-48), las metalo-β-lactamasas, y las cromosómicas de tipo C en P. aeruginosa (AmpC). que son producidas por las súperbacterias más peligrosas en el entorno hospitalario. Hemos desarrollado diversas sulfonas basadas en penicilina que forman aductos estables de indolizidina con el enzima, permitiendo recuperar, tanto in vitro como in vivo, la actividad antibiótica de los carbapenems y las cefalosforinas (Antimicrob. Agents Chemother. 2017, 61, e01172; J. Antimicrob. Chemother. 2016, 7, 2171; Antimicrob. Agents Chemother. 2019, 63, e01092-19). También estamos estudiando la evolución in vivo de la resistencia bacteriana, como fuente de inspiración en el diseño de fármacos (J. Antimicrob. Chemother. 2020, 75, 3209-3217; J. Antimicrob. Chemother. 2021, 76, 91-100).
AGENTES SILENCIADORES – La atenuación de la virulencia bacteriana (Quorum-Quenching) hace que la bacteria pierda su capacidad de causar la infección, y en consecuencia es eliminada por el sistema inmune del huésped. Nos centramos en desactivar ‟Quorum-Quenching‟ el regulador transcripcional LasR de detección del quórum (QS), que es el regulador maestro de QS en P. aeruginosa, que modula la expresión de más de 300 genes (ACS Nano 2015, 9, 5567). También estamos interesados en el diseño de inhibidores inteligentes del enzima dehidroquinasa tipo I (DHQ1), una diana bacteriana anti-virulencia, que actúa como un factor de virulencia in vivo. Hemos desarrollado diversos compuestos funcionalizados con electrófilos poco reactivos, capaces de modificar selectivamente y covalentemente residuos de lisina (Org. Chem. Front. 2019, 6, 3127), y hemos demostrado que DHQ1 cataliza su autoinmolación transformando un grupo 2‐hidroxietilamonio en un epóxido que desencadena la inhibición irreversible (J. Am. Chem. Soc. 2015, 137, 9333; Chem. Eur. J. 2020, 26, 8035).
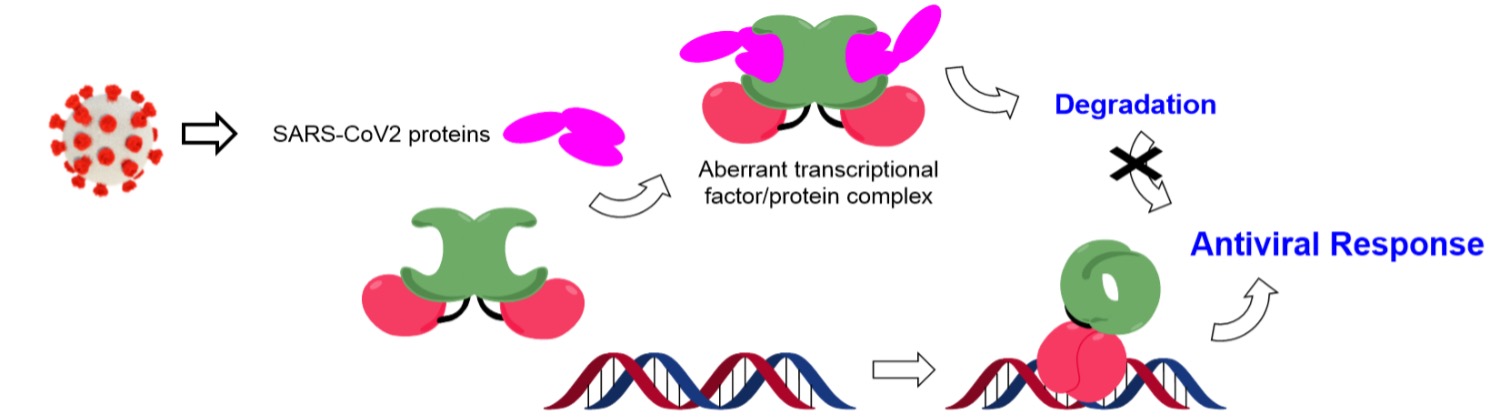
Aproximaciones para Cancelar los Mecanismos de Evasión y Supresión Inmune del SARS-CoV-2
La pandemia de la enfermedad COVID-19 causada por la infección por el nuevo coronavirus 2019 (SARS-CoV-2) se ha convertido en una emergencia de salud pública mundial, cuyo estudio también es el foco de nuestra investigación. La infección por SARS-CoV-2 provoca el trastorno de la inmunidad natural y adaptativa que provoca daño tisular e inflamación sistémica, que es la principal causa de muerte de los pacientes con esta enfermedad. Las proteínas virales a menudo juegan un papel fundamental al interferir con la respuesta inmune del huésped, sin embargo, actualmente se desconoce el mecanismo molecular que subyace a la modulación de las vías de señalización inmune por el SARS-CoV-2. Recientemente nos hemos interesado en explorar el mecanismo molecular detrás de este complejo y desafiante proceso viral, así como en el desarrollo de pequeñas entidades químicas capaces de modularlo. Estamos centrados en el estudio en detalle atómico del mecanismo de activación de la vía de señalización del interferón que establece el estado antiviral, que parece estar modulado por la expresión de pequeñas proteínas capaces de inactivar la producción de interferón en las primeras etapas de la infección. De hecho, la eliminación sistemática de las funciones moduladoras de interferón del virus es un enfoque muy prometedor para el desarrollo de vacunas.
Herramientas para el Diseño
Nuestro grupo de investigación se ha especializado en la aplicación de poderosas herramientas complementarias en el diseño y el descubrimiento de fármacos, como la cristalografía de rayos-X de proteínas, y los estudios computacionales de alto nivel que tienen en cuenta el dinamismo y el mecanismo de acción de la biomacromolécula, simulaciones de Dinámica Molecular, estudios de mecánica cuántica/mecánica molecular (QM/MM). Esto nos permite abordar proyectos más complejos desde una perspectiva multidisciplinar más allá de la química, así como conocer en profundidad los factores clave para la interacción, la inhibición o la modulación de la actividad por parte del ligando. Hemos resuelto más de 30 estructuras cristalográficas de diversos complejos enzima/ligando y aductos, como son los obtenidos con la siquimato quinasa procedente de M. tuberculosis, la deshidroquinasa tipo II procedente de M. tuberculosis y H. pylori o la deshidroquinasa tipo I procedente de S. typhi. y S. aureus. Teniendo en cuenta el movimiento intrínseco del enzima, hemos desarrollado también potentes inhibidores competitivos reversibles con buena actividad in vitro e in vivo frente a H. pylori (Chem. Eur. J. 2016, 22, 17988). Además, empleando simulaciones QM/MM hemos podido determinar el mecanismo catalítico del enzima deshidroquinasa de tipo II (DHQ2), una reconocida diana terapéutica para el descubrimiento de antibióticos, así como identificar los aspectos determinantes de las diferencias de actividad catalítica entre enzimas homólogas de DHQ2 (Org. Biomol. Chem. 2018, 16, 4443), así como el mecanismo de modificación covalente selectiva de enzimas/proteínas, bien inducido por el propio ligando (Chem. Eur. J. 2020, 26, 8035; Org. Chem. Front. 2019, 6, 3127), o por la generación de especies reactivas tras irradiación (J. Org. Chem. 2018, 83, 13019; Chem. Sci. 2017, 8, 2621; Org. Chem. Front. 2019, 6, 99; Chem. Eur. J. 2020, 26, 15922;).




